De novoアセンブリ解析とは
De novoアセンブリ解析とは、ゲノム配列がまだ決定されていない生物種について新規にゲノム配列を決定する解析です。
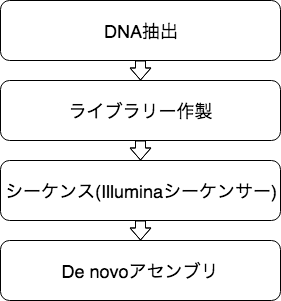
ショートリードによるアセンブリ
De novoアセンブリ解析は、リード配列をつなぎ合わせることでより長い配列を構築する解析です。 より長いリード配列を使用することが望ましいですが、NanoporeやPacBioといったロングリードシーケンサーが出現する以前には、 Illuminaシーケンサー等から得られるショートリードを用いたアセンブリが多く行われていました。 ショートリードを用いたアセンブリのソフトウェアには「SOAPdenovo」、「SPAdes」、「Edena」、「Velvet」等があります。
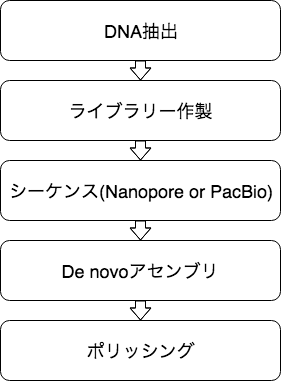
ロングリードによるアセンブリ
NanoporeやPacBioといったシーケンサーの出現により、De novoアセンブリ解析はロングリードシーケンサーを使用して行うことが多くなってきています。 アセンブリには「Canu」、「SMARTdenovo」といったソフトウェアが使われます。 ロングリードのみを用いたアセンブリではエラーが多くなる傾向にあるため、アセンブリの結果についてショートリードを用いてエラー補正することが多く行われます。 これはポリッシングと呼ばれるステップで「Pilon」や「Nanopolish」といったソフトウェアが使われます。