RNAseq解析とは
RNAseq解析とは、NGSを使用してRNAの発現量を定量したり検出を行ったりする解析のことです。 RNAの発現定量には、従来マイクロアレイが多く使われてきましたが、 ダイナミックレンジが狭い、クロスハイブリダイゼーション等の影響を受ける等といった欠点がありました。 RNAseq解析は、これらの欠点を克服することに加えて、新規転写産物を検出できる等といった利点があります。 近年では、Illuminaシーケンサーによるシーケンスの価格が低下していることもあり、マイクロアレイからRNAseqへの置き換えが急速に進んでいます。 第三世代シーケンサーであるNanoporeシーケンサーやPacBioシーケンサーを使用して、完全長の配列を得る手法もありますが、 コストや精度の面で広く普及はしていません。
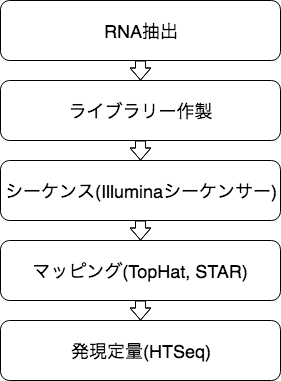
転写産物発現定量
NGSで得られたリードをリファレンス配列にマッピングし、アノテーション情報をもとに転写産物の発現定量を行います。 マッピングには「TopHat」や「STAR」、転写産物の発現定量には「HTSeq」といったソフトウェアが使われます。
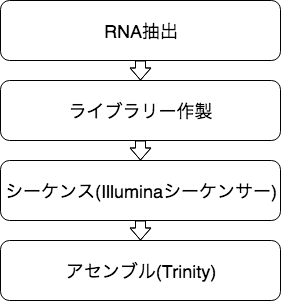
De novo転写産物アセンブリ
リファレンス配列を使用せずに、NGSで得られたリードのみから転写産物の配列をアセンブルします。 「Trinity」といったソフトウェアが使われます。